作者简介:胡广,男,主要从事植物微生物互作研究。E-mail:hugu2012@163.com
为建立一套鉴定棉花miRNA表达丰度的分子技术,优化了Stem-loop RT-PCR方法,并通过分析棉花3个miRNA的表达谱来评价分析该技术体系的特性。首先设计3条引物(茎环特异性反转录引物、正向引物和通用反向引物),通过cDNA和RNA梯度稀释,分析引物的扩增效率和检测灵敏度。结果表明,Stem-loop RT-PCR方法中设计的引物具有较高的扩增效率和检测灵敏度,miR156和miR159扩增效率分别为102.0%和103.6%;并对不同miRNA分析其总RNA检测灵敏度,miR156和miR159的总RNA检测灵敏度差异较大,分别为20 ng和2 pg。同时,应用建立的棉花Stem-loop RT-PCR方法,成功地分析了Gh-miR156、Gh-miR159和Gh-miR5658在根茎叶中的表达量。由此可见,棉花Stem-loop RT-PCR方法具有灵敏度高、特异性强、成本较低和适用于一般实验室等优点,为棉花等植物miRNA相关研究提供了技术保证,加快了miRNA及其调控基因功能的研究步伐。
miRNA functions are involved in plant growth and defense, but simple and highly effective approaches for analyzing miRNA level still need to exploit. In this study, we developed a Stem-loop RT-PCR in cotton to analyze the contents of Gh-miR156, Gh-miR159 and Gh-miR5658. The three special primers were designed, including stem-loop primer, Forward primer and Universal reverse primer. Based on analysis of RT-qPCR Ct values of 10-fold diluted cDNA, the two sets of primers possessed highly amplifying effective for monitoring miR156 and miR159 levels, reached 102.0% and 103.6%, respectively. The sensitivity assays of Stem-loop RT-PCR showed that the limited contents of total RNA for monitoringmiR156 and miR159 were obviously different, which was 20 ng and 2 pg, respectively. Moreover, the expression levels of Gh-miR156, Gh-miR159 and Gh-miR5658 in cotton root, stem and leaf could be well tested by this Stem-loop RT-PCR technique. These results suggested that Stem-loop RT-PCR is well fit for analyzing cotton miRNA level with simple and highly effective potential, which facilitates in plant gene function analysis, especially in miRNA regulation.
microRNAs(miRNAs)是一类只有19~24 nucleotide (nt)的内源性单链非编码小RNA, 广泛存在于真核生物中[1, 2, 3]。研究表明, miRNA在植物代谢[4]、抗病反应[5, 6, 7]、信号传导[8, 9]等方面起着重要的调节作用。由于miRNA序列短小、在生物体中的表达量普遍偏低、同家族之间的miRNA序列差异小等特点, 增加了对miRNA表达量检测的难度[10, 11, 12]。目前, miRNA表达量检测的方法主要依赖于Northern杂交法[13, 14, 15], 该方法虽然可以同时检测多个miRNA的表达情况, 但是其步骤繁琐、操作过程要求严格、涉及到放射性同位素、价格昂贵等, 限制了它的使用[16]。因此, 寻找简单、快速、高效检测miRNA表达量的方法已成为当前的一个研究热点。目前有报道认为, 设计特异性引物的PCR可以有效地检测miRNA表达量, 是当前最灵敏的技术之一[16], 可能成为检测生物miRNA表达量的主要方法。
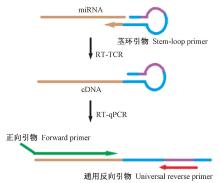
特异引物设计是利用PCR方法检测miRNA的核心技术, 如Poly(A)加尾法是利用Poly(A)聚合酶在miRNA 3'端加一段Poly(A)尾巴对miRNA进行检测[17, 18], 但该方法不能区分同家族的miRNA[19]。PE-qPCR法通过在miRNA 3'端加上一段特异性线性引物(GSP)将miRNA反转录成cDNA, 然后利用一个锁核酸(locked nucleic acid, LNA)修饰的miRNA特异性反向引物和一个与加尾序列一致的通用引物进行PCR扩增[20]。但是线性引物不易区分成熟的miRNA和其前体, 同时需要不断地优化退火温度和反转录的步骤[21]。Stem-loop RT-PCR是通过人为延长miRNA长度以利于miRNA的扩增, 茎环特异性反转录引物由一段通用序列(44 nt)和一段与miRNA的3'端(6 nt)特异性互补的序列组成, 经过退火、反转录后, miRNA被延长为较长的cDNA(图1)[22, 23, 24]。目前, Stem-loop RT-PCR技术已在草莓[25]、番茄[26]等植物中应用。 虽然Stem-loop RT-PCR方法已经呈现出灵敏度高、操作流程简单等优点[27, 28], 但是仍然需要在不同植物中进行条件优化或重建才能广泛地应用于miRNA检测。
近年来, 有一些关于棉花miRNA的相关报道, 如棉花miRNA高通量测序和功能相关分析[29, 30]。这些研究表明棉花miRNA表达水平差异很大, 其中miR156和miR159表达水平较高。miR156和miR159是保守的miRNA家族, 广泛存在于植物中, 调控叶和花的发育[31, 32]。然而, 有关棉花等植物中miRNA表达水平分析的技术方法已经不能满足当前的研究需求, 因此迫切需要开发高效的检测方法来分析miRNA功能。
本试验选择棉花中表达丰度较高的miR156和miR159为研究对象, 探讨Stem-loop RT-PCR方法在棉花中应用。该方法包括设计茎环特异性反转录引物、正向引物、通用反向引物等, 检测miRNA在棉花组织中的表达量, 旨在详细阐述Stem-loop RT-PCR方法在棉花中应用的稳定性、高效性和灵敏性等, 为棉花miRNA的研究提供技术保障。
试验所用材料为中棉35号棉花(Gossypium hirsutum L.), 由山西省农业科学院棉花研究所提供。Pure Link Plant RNA Reagent (TRIzol)购自美国Invitrogen公司; 反转录试剂盒和RT-qPCR试剂盒等均购自北京全式金公司; 引物合成由北京三博远志生物技术有限公司完成。
1.2.1 引物序列设计 由于miRNA片段较短且没有poly(A)尾巴, 不能按常规方法进行反转录和PCR扩增, 因此设计茎环特异性反转录引物对miRNA进行特异性反转录(图1)。在miRNA 数据库miRbase (http://www.mirbase.org/)中下载miR156(登录号:MI0005638)、miR159(登录号:MIMAT0000177)、miR5658(登录号:MI0019242)的序列, 然后设计3条引物:一条茎环特异性反转录引物(stem-loop primer)用于RT-PCR, 一条正向引物(forward primer)用于PCR扩增, 一条通用反向引物(universal reverse primer)用于半定量RT-PCR和实时荧光定量PCR(RT-qPCR)。在每个茎环特异性反转录引物的内部都设计一段固定的序列, 且能形成一个茎环结构, 其5'端共有44个固定核苷酸序列(5'-G T C G T A T C C A G T G C A G G G T C C G A G G T A T T C G C A C T G G A T A C G AC-3'), 在固定核苷酸序列的5'端(5'-G T C G T A T C C A G T GC-3')和3'端(5'-G C A C T G G A T A C G AC-3')反向互补构成长14 bp的茎结构, 余下的16 nt构成茎环引物的环结构; 在茎环引物3'端设计6 nt与miRNA的3'端反向互补。正向引物长约17~24 nt, 在3'端有13~18 nt与miRNA的5'端互补。在正向引物的5'端加4~6 nt保护碱基, 使退火温度控制在60℃左右。通用的反向引物长16 nt, 位于茎环引物5'端的第11~第26位碱基上, 引物序列见表1。
1.2.2 TRIzol法提取总RNA 称取棉花的组织样品100 mg, 用液氮研磨至粉末状, 根据Pure Link Plant RNA Reagent的使用说明, 采用TRIzol 法提取总RNA。加入10~30 μ L RNase-free Water溶解总RNA, 总RNA样品用1.5%琼脂糖电泳检测RNA完整性, NanoDrop ND-2000 分光光度计(NanoDrop Technologies, Wilmington, DE, USA)测定RNA的浓度。
1.2.3 Stem-loop RT-PCR 试验使用反转录试剂盒合成cDNA。具体操作:于冰上配制反应体系(20 μ L), 加入1 μ L stem-loop primer(1 μ mol· L-1)、RNase-free Water和RNA(1 μ g), 将混合物短暂离心, 65℃金属浴5 min, 冰浴2 min。加入2× TS Reaction Mix (10 μ L), TransScript RT/RI Enzyme Mix (1 μ L), gDNA Remover (1 μ L), 轻轻混均, 短暂离心。采用脉冲逆转录反应进行cDNA第一链的合成, 具体程序如下:首先16℃孵育30 min, 然后进行60个循环的脉冲反应(每个循环:30℃ 30s, 42℃ 30s, 50℃ 30s), 最后反应液进行85℃ 5s灭活反转录酶。反转录得到的cDNA分装-20℃贮存在, 将反转录后的cDNA稀释10倍后使用。U6基因使用随机引物(random primer)反转录, 25℃孵育10 min, 42℃孵育30 min, 最后反应液进行85℃ 5s灭活反转录酶。为了检测Stem-loop RT-PCR的灵敏度, 对RNA按10倍梯度稀释, 终浓度分别是2 pg、20 pg、200 pg、2 ng、20 ng、200 ng, 进行逆转录反应, 合成cDNA后备用。
| 表1 miRNA引物和序列 Table 1 The miRNA sequences and primers |
1.2.4 半定量 RT-PCR以反转录后稀释10倍的cDNA为模板, 加入相应引物(表1)进行半定量RT-PCR反应。于冰上配制反应体系(20 μ L):10× PCR buffer (2 μ L)、dNTP Mixture (0.4 μ L)、forward primer (0.4 μ L)、universal reverse primer (0.4 μ L)、cDNA (2.0 μ L)、Easy Taq(0.1 μ L)、ddH2O (14.7 μ L); PCR程序:94℃预变性2 min, 进行35个循环扩增, 94℃变性15 s, 58℃退火和延伸1 min, 4℃保存, 使用4%琼脂糖凝胶电泳检测扩增结果。
1.2.5 RT-qPCR 试验使用全式金提供的反应试剂盒(TransStart Top Green qPCR SuperMix Kit)、于冰上避光配制反应体系(20 μ L):qPCR SuperMix (10 μ L)、forward primer (0.4 μ L)、universal reverse primer (0.4 μ L)、ddH2O(6.2 μ L)。轻轻混均, 短暂离心。将 18 μ L 不含cDNA模板的混合物加入RT-qPCR专用96孔板中, 然后每孔加入稀释10倍的cDNA 2 μ L。RT-qPCR程序:95℃预变性5 min, 45个循环, 95℃变性 5 s, 60℃退火10 s, 72℃延伸1 s。熔解曲线程序:从65℃加热至95℃, 每秒升高0.5℃, 确定扩增产物的特异性。每个样品作3次重复, U6作为内参基因(NCBI登录号:XM_016853274), 2-△ △ Ct法[33]计算相对表达量。
1.2.6 扩增效率的计算 以10倍梯度稀释的cDNA模板, 进行RT-qPCR分析。以稀释倍数为横坐标, Ct值为纵坐标制作标准曲线。通过公式E=(10(-1/s)-1)× 100%计算引物的扩增效率, 其中S为标准曲线斜率[33]。
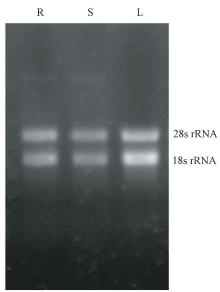
分析棉花miRNA表达量时, 对其总RNA质量存在着较高的要求, 因此, 首先对TRIzol试剂提取的棉花总RNA质量进行分析。由图2可知, 总RNA经1.5%的琼脂糖凝胶电泳检测, 28s rRNA和18s rRNA条带亮度清晰, 没有拖带、杂带, 表明提取的总RNA的完整性较好; 棉花样品的总RNA浓度在500~950 ng· μ L-1, OD260/OD280值在1.9~2.1之间, 说明提取的棉花总RNA质量较好, 能够保证后续试验的进行。
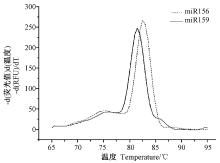
为了评价Stem-loop RT-PCR在棉花中应用, 必须先对设计的引物扩增效率进行检验。先利用茎环特异性反转录引物将RNA反转录成cDNA, 对cDNA进行10倍梯度的稀释, 进行RT-qPCR。对获得的Ct值和稀释梯度进行相关性分析。由图3可知, miR156和miR159扩增的Ct值和稀释梯度成显著正相关关系, 相关系数R2均为0.999, 其回归方程的斜率分别为SmiR156=3.27和SmiR159=3.24, 由此计算出miR156 和miR159的PCR扩增效率分别为102.0%和103.6%。结果表明, 在10-4~10梯度稀释cDNA的拷贝数范围内引物扩增效率高。此外, RT-qPCR产物的融解曲线, 在82℃左右出现单一峰, 无杂峰, 进一步证明了引物具有良好的特异性(图4)。总之, 本研究设计的茎环特异性反转录引物、正向引物、通用反向引物可以用于棉花中miRNA的PCR检测分析, 能准确地反映模板中miRNA的相对表达量。
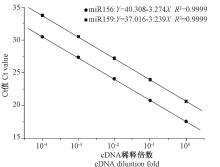 | 图3 miR156、miR159引物扩增标准曲线Fig.3 Standard curve graph of amplification of miR156 and miR159 with the specific primers |
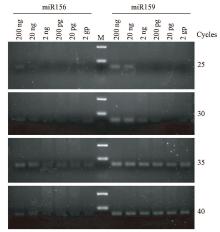
在使用Northern blot分析miRNA表达量时, 一般需要2 μ g植物RNA[22]。为确立Stem-loop RT-PCR方法的检测灵敏度, 将提取的棉花叶组织的总RNA作一系列梯度稀释, miR156和miR159在茎环特异性反转录引物特异性反转录后, 直接进行半定量RT-PCR分析。结果表明, Stem-loop RT-PCR扩增35个循环能检测到2 pg的棉花总RNA中的miR159, 而对于棉花miR156表达量的检测, 则至少需要20 ng的总RNA, 且扩增循环数在35个以上(图5)。同时, 在没有加茎环特异性反转录引物反转录的cDNA中没有扩增出特异条带(数据略)。此外, 所有扩增反应中均未发现引物二聚体, 也说明Stem-loop RT-PCR方法在棉花miRNA表达量分析具有较高的特异性。
为了进一步确定Stem-loop RT-PCR在棉花miRNA表达量检测中的高效性, 选择表达量高的Gh-miR156, Gh-miR159和表达量低的Gh-miR5658进行组织特异性分析。结果表明, Stem-loop RT-PCR方法可以有效地检测3个miRNA在棉花根、茎、叶中的表达量。由图6可知, Gh-miR159在棉花的根、茎、叶中丰度最高, Gh-miR156在棉花的根、茎、叶中丰度也比较高, 而Gh-miR5658在根、茎、叶中的丰度相对很低。同时, 这3个miRNA在根茎叶中的表达量存在明显差异, 暗示它们可能在不同的组织中有特异的功能。这些结果进一步说明, Stem-loop RT-PCR方法适用于棉花不同表达量的miRNA定量分析。本研究建立的Stem-loop RT-PCR具有灵敏度高、特异性强、成本较低、操作流程简单等优点, 可以普遍地应用在棉花miRNA表达量检测。
植物miRNA丰度的检测经典的方法就是利用分子杂交, 但是该方法涉及到放射性同位素, 且技术繁杂和成本高等缺点, 同时对丰度低的miRNA难以适用。近年来, 有些报道集中于利用特异引物设计的PCR方法来检测植物miRNA丰度[27, 28], 极大地推动了植物miRNA的研究。本研究在棉花中建立Stem-loop RT-PCR方法, 利用这个技术可以快速、方便地检测出不同表达丰度的棉花miRNA, 为棉花miRNA的研究提供前提条件。
利用Stem-loop RT-PCR进行miRNA丰度检测已有一些报道, 但是这些报道往往是集中在miRNA尾部加poly(A)结构或者是加基因特异性引物(GSP), 加poly(A)的多用于测序和高通量数据分析[17]。在miRNA尾部加GSP的方法虽然可以检测miRNA的丰度, 但是很难区分前体miRNA的干扰[20]。因此, 仅有少量报道Stem-loop RT-PCR方法的研究, 主要在草莓[25]、番茄[26]中进行miRNA丰度的分析。Stem-loop RT-PCR在棉花等大田作物miRNA表达丰度检测中的应用尚不多见。本研究设计的茎环特异性反转录引物能够很好的于miRNA表达量的检测分析, 分别利用半定量RT-PCR和RT-qPCR来验证这个miRNA表达量检测体系, 通过引物检测效率和不同miRNA检测灵敏度分析, 证明建立的Stem-loop RT-PCR体系适合棉花不同miRNA的丰度分析。
利用分子杂交技术检测植物miRNA表达水平是一种经典的方法, 但该方法不能够分析低水平表达的miRNA, Wang等[34]在拟南芥中用分子杂交方法未能检测到miR847, 但是在测序时发现miR847为低丰度表达。本研究中的Stem-loop RT-PCR方法不仅成功地分析了高丰度Gh-miR156和Gh-miR159的表达谱, 也能检测出低丰度Gh-miR5658的组织特异性表达。在草莓[25]和番茄[26]中也有相似的报道。因此Stem-loop RT-PCR方法对于低丰度miRNA的检测具有较高的灵敏度。
本研究建立的棉花Stem-loop RT-PCR技术具有灵敏度高、特异性强、成本较低、操作流程简单等优点。引物扩增效率分析结果表明, Gh-miR156和Gh-miR159的扩增效率分别为102.0%和103.6%; RT-qPCR中的Ct值相差标准值分别为3.27和3.24, 一般浓度相差10倍的cDNA在RT-qPCR中的Ct值相差标准值为3.32, 该试验数值略小于标准值, 与理论值差异不显著, 表明在棉花中设计的茎环特异性反转录引物具有高的扩增效率。然而, 祁心等[26]在蕃茄中设计的茎环特异性反转录引物的扩增效率偏离标准值较大, 认为可能是试验操作不精细造成的误差。
本研究建立并优化了一套检测棉花miRNA表达丰度的方法— — Stem-loop RT-PCR。设计不同的特异茎环结构引物, 利用Stem-loop RT-PCR检测出棉花3个miRNA的不同表达丰度, 包括在根、茎、叶中表达水平。同时分析设计引物的扩增效率和不同miRNA的检测总RNA水平阈值。结果表明, 本研究建立的Stem-loop RT-PCR是一套简单、高效检测棉花miRNA表达量的方法, 该方法的建立可能推动棉花miRNA的深入研究。
The authors have declared that no competing interests exist.
| [1] |
|
| [2] |
|
| [3] |
|
| [4] |
|
| [5] |
|
| [6] |
|
| [7] |
|
| [8] |
|
| [9] |
|
| [10] |
|
| [11] |
|
| [12] |
|
| [13] |
|
| [14] |
|
| [15] |
|
| [16] |
|
| [17] |
|
| [18] |
|
| [19] |
|
| [20] |
|
| [21] |
|
| [22] |
|
| [23] |
|
| [24] |
|
| [25] |
|
| [26] |
|
| [27] |
|
| [28] |
|
| [29] |
|
| [30] |
|
| [31] |
|
| [32] |
|
| [33] |
|
| [34] |
|